RnaSecondaryStructurePrediction에서 DynamicProgramming에 기반한 [RNA] 2차구조 예측 [Algorithm]. 정확한 예측은 무리. 그러나 짧은 [RNA]에 대해서는 비교적 정확한 결과를 제공한다.
본 계산은 [Recursion]을 이용한다. 짧은 서열에 대해 최적구조를 계산하고, 이를 크게 확장시켜나간다. 재귀의 키포인트는 짧은 서열에서의 최적구조 i, j는 다음의 4가지 종류만이 있다는 점이다.
- add unpaired position i onto bast structure for subsequence i+1, j
- add unpaired position j onto best structure for subsequence i, j-1
- add i, j pair onto best structure found for subsequence i+1, j-1
- combine two optimal substructures i, k and k+1, j
길이 L의 서열 x 
에서 base pairing 이 일어나는 위치는 구하는 알고리즘은 다음의 두 단계로 이루어진다.
Fill stage
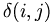
를 정의하고 i, j가 complementary base pair이면 1, 아니면 0을 준다. 그리고는 점수 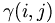
를 다음처럼 구한다.
Initialisation 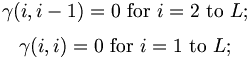
Recursion: starting with all subsequences of length 2, to length L: ![$$ \gamma(i,j) = \textrm{max} \left \{ \begin{array}{ll}
\gamma(i+1,j), \\
\gamma(i, j-1), \\
\gamma(i+1, j-1) + \delta(i,j), \\
\textrm{max}_{i<k<j} \biggl[ \gamma(i,k) +\gamma(k+1,j) \biggl]
\end{array} \right $$ $$ \gamma(i,j) = \textrm{max} \left \{ \begin{array}{ll}
\gamma(i+1,j), \\
\gamma(i, j-1), \\
\gamma(i+1, j-1) + \delta(i,j), \\
\textrm{max}_{i<k<j} \biggl[ \gamma(i,k) +\gamma(k+1,j) \biggl]
\end{array} \right $$](/wiki/NussinovRnaFoldingAlgorithm?action=AttachFile&do=get&target=c3ff2d120817f4c65f5a8af4b405e52bb4b280b6_latex.png)
Traceback stage
trace back은 Stack을 써서 다음처럼 한다.
Initialisation
Push(l,L) onto stack.
Recursion: Repeat until stack is empty:
- pop (i,j)
- if i >= j continue;
else if gamma(i+1,j) = gamma(i,j), push (i+1, j);
else if gamma(i,j-1) = gamma(i,j), push (i, j-1);
else if gamma(i+1, j-1) + delta(i,j) = gamma(i,j):
- record i, j base pair.
- push (i+1, j-1).
else for k = i+1 to j-1: if gamma(i,k) + gamma(k+1, j) = gamma(i,j):
- push (k+1, j).
- push (i,k).
- break.PythonCode: [Nussinov.py], UnitTest code [TestNussinov.py]
(2001-12-11 현재 완료하긴 했는데 교재랑 결과값이 다르게 나옵니다. 책대로라면, 서열 GGGAAAUCC 에 대해 [(2,9),(3,8),(4,7)]가 나와야하는데 [(2,9),(3,8),(6,7)]이 출력됩니다. 책 그림을 봐도 마지막부분에 1에서 1로 가야하는데 0 으로 가버리네요)
See also ScfgNussinovRnaFoldingAlgorithm (StochasticContextFreeGrammar-based algorithm)
 BioHackersNet
BioHackersNet