|
Size: 1603
Comment:
|
Size: 1609
Comment:
|
| Deletions are marked like this. | Additions are marked like this. |
| Line 8: | Line 8: |
| T_m = 4(G+C) + 2(A+T) | $$ T_m = 4(G+C) + 2(A+T) $$ |
[Primer] [Design]. [PCR]을 통해 특정서열을 증폭해내고자 할때, 원하는 서열을 콕 집어내려면, 서열을 보고 적절한 primer를 디자인해야한다.
약 18-24mer의 oligomer서열과 Tm값을 계산해낼 수 있다. annealing온도의 몇도차이가, 현저한 [PCR]결과를 만들어낸다.
20base이내인 primer의 Tm값은 다음공식으로 계산될 수 있다.
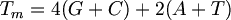
그 이상의 길이에 대해서는 NearestNeighobr계산이 필요하다.
Primer내에 적당한 GC contents가 필요하다. 이 GC함량과 Tm과 값을 잘 맞추어야한다.
[PCR] 생성물의 길이는 PCR 증폭시 그 효율성에 상당히 영향을 준다. 생성물의 길이는 그 목적에 따라 달라지는데, 특정 부위를 확인하는 목적이라면 120-300 bp가 적당하다.
관련정보
관련프로그램
- [Primer3]
[PrimerDesigner.py] : [Primer3]을 이용한 스크립트
- [Amplicon]
궁금함중 한가지. m[RNA]의 5'UTR영역이 [Gene] specific하기에, 그부분으로 파악되는 부분으로 PrimerDesign을 하면 좋다고~~~ 왜, 5'UTR영역이 Gene specific한가? 이거, 분자생물학책에 나오는건데 내가 모르고 있는건가? AnswerMe~~ --[yong27/2004-08]
 BioHackersNet
BioHackersNet