|
Size: 1587
Comment: converted to 1.6 markup
|
Size: 1650
Comment:
|
| Deletions are marked like this. | Additions are marked like this. |
| Line 8: | Line 8: |
| * 반복염기서열 수의 차이 : Satellite, Minisatellite, [Microsatellite] ([STR]) | * 반복염기서열 수의 차이 : Satellite, Minisatellite, [[Microsatellite]] ([[STR]]) |
| Line 11: | Line 11: |
| * [SNP] : 전체 인간 유전변이형의 90% 이상 ( > 11,000,000 SNPs (290 bp 당 1개)) | * [[SNP]]: 전체 인간 유전변이형의 90% 이상 ( > 11,000,000 SNPs (290 bp 당 1개)) * [[CNV]]: 좀 더 큰 영역의 삽입 혹은 삭제 |
| Line 42: | Line 43: |
| 1. [질병유전체분석법] | 1. [[질병유전체분석법]] |
유전적변이
유전적변이
- 돌연변이는 평균적으로 염기서열 당 약 2.5 x 10^(-8)의 확률로 발생한다.
- 이는 한 세대에서 약 175개의 돌연변이를 만든다.
유전적변이의 종류
반복염기서열 수의 차이 : Satellite, Minisatellite, Microsatellite (STR)
산발적으로 존재하는 반복 DNA RepetitiveSequence :LINE, SINE, transposable elements
- 삽입과 결손 : indel
SNP: 전체 인간 유전변이형의 90% 이상 ( > 11,000,000 SNPs (290 bp 당 1개))
CNV: 좀 더 큰 영역의 삽입 혹은 삭제
Microsatellite와 SNP의 비교
Microsatellite |
SNPs |
많은 대립유전자형을 가지고 있어서 분별력이 높음 |
대부분 2개의 bi-alleles, 분별력이 떨어짐 |
유전적 전달의 정확성이 약간 떨어짐 (마커의 반복 수 팽창 및 높은 돌연변이율) |
유전적 전달의 정확성이 매우 높음 |
인간유전체에 약 50,000개 존재 |
11,000,000개 이상 존재 1 SNP / 290 bp) |
최초의 유전지도 작성에 이용 |
연관성 연구에 사용 |
다양성의 측정
segregatting site 개수 k (길이 L 당 개수 K)
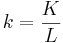
nucleotide diversity pi
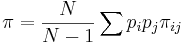
집단간 유전적차이 FST
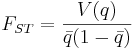
- V(q) = A2 유전자형 빈도의 분산값
- q = sum(wi qi) 로 A2 유전자형의 weighted frequency
 BioHackersNet
BioHackersNet