|
Size: 951
Comment:
|
← Revision 22 as of 2013-07-12 17:16:19 ⇥
Size: 1437
Comment:
|
| Deletions are marked like this. | Additions are marked like this. |
| Line 2: | Line 2: |
base calling : 크로마토그램의 피크를 분석하여 염기서열을 할당하는 것 |
|
| Line 13: | Line 15: |
| \math{ QV $=-10 \log{P_e} } |
$$ QV = -10 \log{P_e} $$ |
| Line 18: | Line 18: |
| QV가 40정도는 되어야 시퀀싱 정확도가 99.99%가 된다. | Quality value : '''QV''' , base call error probability : '''Pe''' QV가 40정도는 되어야 시퀀싱 정확도가 99.99%가 된다. (QV 30 : 99.9% , QV 20 : 99%) |
| Line 25: | Line 27: |
| 최근 [SNP] detection을 위한 PolyPhred가 소개되었다. | phred 프로그램의 옵션중에 QV가 낮은 영역을 trim하는 것이 있다. assembly할 목적이라면, 이 옵션은 켜지 않는것이 좋다. 최근 [[SNP]] detection을 위한 PolyPhred가 소개되었다. [[http://210.115.163.141/study/bioinformatics/PhredPhrep/index.htm|phred/phrap/consed 슬라이드]] |
| Line 28: | Line 34: |
---- 질문있어요~~~ |
Base calling 프로그램. Sequencing 머신이 만들어낸 이미지파일(ABI, SCF, ESD등)을 읽어서 FastaFormat으로 변환하는 프로그램이다. 그래프이미지내의 Chromatogram의 곡선을 보고 적당한 Base를 지정함과 동시에 score까지 만들어서 파일로 만든다. FourierTransform [Algorithm]이 사용되었다.
base calling : 크로마토그램의 피크를 분석하여 염기서열을 할당하는 것
작동가능한 시퀀싱머신
- ABI 373,377,3700
- Molecular Dynamics MegaBACE
- LI-COR 4000
Phred가 만들어내는 score를 QV (Quality Value)라고 한다.
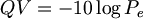
Quality value : QV , base call error probability : Pe
QV가 40정도는 되어야 시퀀싱 정확도가 99.99%가 된다. (QV 30 : 99.9% , QV 20 : 99%)
간단히 다음처럼 사용할 수 있다. 추가적인 사용법은 LargeScaleSequencing페이지 참고.
$ phred -id chromat_dir -sa seqs_fasta -qa seqs_fasta.qual
phred 프로그램의 옵션중에 QV가 낮은 영역을 trim하는 것이 있다. assembly할 목적이라면, 이 옵션은 켜지 않는것이 좋다.
최근 SNP detection을 위한 PolyPhred가 소개되었다.
질문있어요~~~
 BioHackersNet
BioHackersNet