|
Size: 4099
Comment:
|
Size: 4113
Comment:
|
| Deletions are marked like this. | Additions are marked like this. |
| Line 56: | Line 56: |
| {{{ $$ P(k|DI) \propto P(k|I)P(D|kI) = P(k|I) \frac{(N-r)!}{N!} \prod_{i=i}^{r} \bigg\{ \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min}}}{\sigma_i} \times \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ |
{{{#!latex $$ P(k|DI) \propto P(k|I)P(D|kI) = P(k|I) \frac{(N-r)!}{N!} \prod_{i=i}^{r} \bigg\{ \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{\sigma_i} \times \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ |
| Line 62: | Line 62: |
| {{{#! $$ P(k|DI) \sim P(k|I) \bigg( \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min}}}{N} \bigg)^r \times \prod_{i=i}^{r} \frac{1}{\sigma_i} \bigg\{ \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ |
{{{#!latex $$ P(k|DI) \sim P(k|I) \bigg( \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{N} \bigg)^r \times \prod_{i=i}^{r} \frac{1}{\sigma_i} \bigg\{ \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ |
Annl.Chem. 2000, 72, 2482-2489, 10857624
ProFound : An ExpertSystem for ProteinIdentification Using MassSpectrometry Peptide Mapping Information
http://prowl.rockefeller.edu/zhangw/ac991363o.pdf
Introduction
기존의 PeptideMassFingerprint 방법
- number of matches
- [MOWSE] score
[MOWSE]점수는 각 [Protein]의 개별성질들을 반영하지 못한다. 따라서 본 논문에서는 BayesianInference로 개별성질들을 반영한 점수화방법을 제안한다. 여기서의 개별성질들은
peptide mass
- mass range
- species
- mass accuracy
- [Enzyme] cleavage chemistry
- [Protein] sequence
- previous experiments on the sample [Protein]
AminoAcid contents
Method
designate
- k : protein entry in [Database]
- D : experimental data
I : background information (PriorKnowledge such as
- species,
- approximate mass,
- mass accuracy,
- [Enzyme] cleavage chemistry,
- previous experiments on the sample protein)
assumption
- 그 단백질이 DB에 존재한다.
- 모든 관측된 질량값들은 그 단백질에서 나온것이다.
- 실험치 질량값과 이론치 질량값의 매치는 그 단백질이기때문에 일어나는것이다. (random match제외)
P(k|DI)를 구한다. BayesRule에 의해서
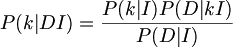
여기에서
P(k|I) : 해당 background정보인 PriorProbability
- P(D|kI) : 가설이 맞을경우 데이터 D가 관측될 Likelihood probability
- P(D|I) : k와 관계없는 상수
따라서, 다음처럼 정리할 수 있다.
![$$ P(k|DI) \propto P(k|I)P(D|kI) = P(k|I) \frac{(N-r)!}{N!} \prod_{i=i}^{r} \bigg\{ \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{\sigma_i} \times \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ $$ P(k|DI) \propto P(k|I)P(D|kI) = P(k|I) \frac{(N-r)!}{N!} \prod_{i=i}^{r} \bigg\{ \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{\sigma_i} \times \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$](/wiki/ProFoundAnExpertSystemForProteinIdentification?action=AttachFile&do=get&target=a33efffa2b1555dfe247b3f101fef3e8c7cda84a_latex.png)
위식은, N이 커질경우, 다음식에 수렴한다.
![$$ P(k|DI) \sim P(k|I) \bigg( \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{N} \bigg)^r \times \prod_{i=i}^{r} \frac{1}{\sigma_i} \bigg\{ \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$ $$ P(k|DI) \sim P(k|I) \bigg( \sqrt{\frac{2}{\pi}} \frac{m_{\mathrm{max}} - m_{\mathrm{min} }}{N} \bigg)^r \times \prod_{i=i}^{r} \frac{1}{\sigma_i} \bigg\{ \sum_{j=1}^{g_i} \exp \bigg[ - \frac{(m_i - m_{ij0})^2}{2\sigma_i^2} \bigg] \bigg\} F_{\mathrm{pattern}} $$](/wiki/ProFoundAnExpertSystemForProteinIdentification?action=AttachFile&do=get&target=cc83ba171b25733a9dbb6b74232648474898e161_latex.png)
위식을 통해 다음을 알 수 있다. [Database]내에 주어진 단백질 k에 대해, k가 sample 단백질일 확률은 number of hit r에 따라 증가하고, mass accuracy(i.e. smaller sigma, mi-mij0)에 따라 증가하고, 이론치 조각갯수 N에 따라 감소한다.
tag information : 주어진 peptide에서 알고있는 특별한 AminoAcid들.
- cys : chemical alkylation of free thiol moiety
- met : 16 Da 차이로 쪼개짐 (partially oxidized)
empirical factor : 이경우 확률은 증가된다.
- adjacency
- common-end overlapping
current ProFound input parameter
- taxonomy category
- mass range
- digestion chemistry
- maximum number of missed cleavage sites (digestion정도에 따라 조절)
- modification
Result and Discussion
Identification of Single Isolated Proteins
RS4B_SCHPO 을 예제로 설명. 사용한 parameter는
Saccharomyces cerevisiae
- 35 monoisotopic masses
- mass range 0-3000 kDa
- unmodified cystenis
- maximum missed cleavage sites 2
- mass tolerance 0.1 Da
23개의 실험치질량들이 24개의 이론치질량과 매치. 70% coverage sequence. scatter plot으로 error표시(systematic error의 시각화)
CH60_HUMAN 을 예제한 설명에서는 mouse,rat,hamster에서 homologous protein이 바로 다음 후보로 나왔다.
Identification of Protein Components in mixture
binary mixture로 검색.
Independent verification of the PeptideMassFingerprint
TandemMassSpectrometry 를 이용해서 verification
Improvement of the confidence level using tag information
cys가 있느냐 없느냐에 관한 정보가 ProteinIdentification의 확신을 증가시킴
 BioHackersNet
BioHackersNet